작은 분자 그래프 생성을 위한 R-GCN이 포함된 WGAN-GP
- 원본 링크 : https://keras.io/examples/generative/wgan-graphs/
- 최종 확인 : 2024-11-23
저자 : akensert
생성일 : 2021/06/30
최종 편집일 : 2021/06/30
설명 : 새로운 분자를 생성하기 위해 R-GCN을 갖춘 WGAN-GP의 완전한 구현.
소개
이 튜토리얼에서는, 그래프 생성 모델을 구현하고 이를 사용하여 새로운 분자를 생성합니다.
동기: 신약 개발 (분자 설계)은 시간이 많이 걸리고 비용이 많이 듭니다. 딥러닝 모델을 사용하면, 알려진 분자의 속성(예: 용해도, 독성, 표적 단백질에 대한 친화도 등)을 예측하여, 적합한 후보 약물을 찾는 과정을 단축할 수 있습니다. 가능한 분자의 수는 천문학적이므로 우리가 탐색하는 분자 공간은 전체 공간의 일부에 불과합니다. 따라서, 새로운 분자를 생성할 수 있는 생성 모델을 구현하는 것이 바람직하다고 할 수 있습니다. (이전에는 탐색되지 않았을 가능성이 큽니다)
참고문헌 (구현)
이 튜토리얼의 구현은 MolGAN 논문과 DeepChem의 Basic MolGAN을 기반으로 하고 있습니다.
추가 읽기 (생성 모델)
최근 분자 그래프를 위한 생성 모델 구현으로는
등이 있습니다.
생성적 적대 신경망에 대한 추가 정보는
를 참조하세요.
셋업
RDKit 설치
RDKit은 C++ 및 Python으로 작성된 화학정보학 및 머신러닝 소프트웨어 모음입니다. 이 튜토리얼에서는 SMILES를 분자 객체로 변환하고, 해당 객체로부터 원자와 결합 세트를 얻는 데 RDKit을 사용합니다.
SMILES는 주어진 분자의 구조를 ASCII 문자열 형태로 표현합니다. SMILES 문자열은 더 작은 분자에 대해 상대적으로 사람이 읽기 쉬운 간결한 인코딩을 제공합니다. 분자를 문자열로 인코딩하면 데이터베이스 및/또는 웹에서 분자를 검색할 때 매우 편리합니다. RDKit은 주어진 SMILES를 분자 객체로 정확하게 변환하는 알고리즘을 사용하며, 이를 통해 많은 분자 속성/특징을 계산할 수 있습니다.
RDKit은 보통 Conda를 통해 설치됩니다. 그러나 rdkit_platform_wheels 덕분에, 이 튜토리얼에서는 pip를 통해 쉽게 설치할 수 있습니다.
pip -q install rdkit-pypi분자 객체를 쉽게 시각화하려면, Pillow도 설치해야 합니다.
pip -q install Pillow패키지 Import
from rdkit import Chem, RDLogger
from rdkit.Chem.Draw import IPythonConsole, MolsToGridImage
import numpy as np
import tensorflow as tf
from tensorflow import keras
RDLogger.DisableLog("rdApp.*")데이터세트
이 튜토리얼에서 사용된 데이터셋은 MoleculeNet에서 얻은, 양자 역학 데이터셋 (QM9)입니다. 이 데이터셋에는 많은 특성과 레이블이 포함되어 있지만, 우리는 주로 SMILES 열에 집중할 것입니다. QM9 데이터셋은 그래프 생성을 위한 첫 번째 작업으로 적합한 데이터셋입니다. 그 이유는 분자에서 발견되는 최대 비수소 원자 수가 9개에 불과하기 때문입니다.
csv_path = tf.keras.utils.get_file(
"qm9.csv", "https://deepchemdata.s3-us-west-1.amazonaws.com/datasets/qm9.csv"
)
data = []
with open(csv_path, "r") as f:
for line in f.readlines()[1:]:
data.append(line.split(",")[1])
# 데이터셋의 분자 중 하나를 살펴보겠습니다.
smiles = data[1000]
print("SMILES:", smiles)
molecule = Chem.MolFromSmiles(smiles)
print("Num heavy atoms:", molecule.GetNumHeavyAtoms())
molecule결과
SMILES: Cn1cncc1O
Num heavy atoms: 7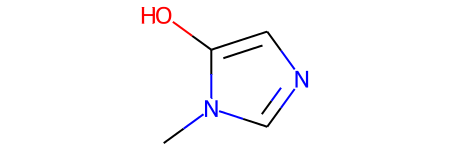
보조 함수 정의
이 보조 함수들은 SMILES를 그래프로 변환하고 그래프를 분자 객체로 변환하는 데 도움을 줍니다.
분자 그래프 표현. 분자는 자연스럽게 G = (V, E)와 같은 무방향 그래프로 표현될 수 있습니다.
여기서 V는 정점, vertices(원자, atoms)의 집합이며, E는 간선, edges(결합, bonds)의 집합입니다.
이 구현에서는, 각 그래프(분자)가 원자 쌍의 존재/부재를 인코딩한 인접 텐서 A와
각 원자의 원자 유형을 원-핫 인코딩한 특징 텐서 H로 표현됩니다.
여기서 수소 원자는 RDKit을 통해 추론할 수 있으므로,
모델링을 쉽게 하기 위해 A와 H에서 수소 원자는 제외됩니다.
atom_mapping = {
"C": 0,
0: "C",
"N": 1,
1: "N",
"O": 2,
2: "O",
"F": 3,
3: "F",
}
bond_mapping = {
"SINGLE": 0,
0: Chem.BondType.SINGLE,
"DOUBLE": 1,
1: Chem.BondType.DOUBLE,
"TRIPLE": 2,
2: Chem.BondType.TRIPLE,
"AROMATIC": 3,
3: Chem.BondType.AROMATIC,
}
NUM_ATOMS = 9 # 최대 원자 수
ATOM_DIM = 4 + 1 # 원자 유형의 수
BOND_DIM = 4 + 1 # 결합 유형의 수
LATENT_DIM = 64 # 잠재 공간의 크기
def smiles_to_graph(smiles):
# SMILES를 분자 객체로 변환
molecule = Chem.MolFromSmiles(smiles)
# 인접 텐서와 특징 텐서 초기화
adjacency = np.zeros((BOND_DIM, NUM_ATOMS, NUM_ATOMS), "float32")
features = np.zeros((NUM_ATOMS, ATOM_DIM), "float32")
# 분자에서 각 원자에 걸쳐 반복
for atom in molecule.GetAtoms():
i = atom.GetIdx()
atom_type = atom_mapping[atom.GetSymbol()]
features[i] = np.eye(ATOM_DIM)[atom_type]
# 원-홉 이웃을 반복
for neighbor in atom.GetNeighbors():
j = neighbor.GetIdx()
bond = molecule.GetBondBetweenAtoms(i, j)
bond_type_idx = bond_mapping[bond.GetBondType().name]
adjacency[bond_type_idx, [i, j], [j, i]] = 1
# 결합이 없는 경우, 마지막 채널에 1을 추가 (결합 없음 표시)
# 주의: 채널이 처음 (channels-first)
adjacency[-1, np.sum(adjacency, axis=0) == 0] = 1
# 원자가 없는 경우, 마지막 열에 1을 추가 (원자 없음 표시)
features[np.where(np.sum(features, axis=1) == 0)[0], -1] = 1
return adjacency, features
def graph_to_molecule(graph):
# 그래프 언팩
adjacency, features = graph
# RWMol은 편집을 위한 분자 객체입니다
molecule = Chem.RWMol()
# "원자가 없는" 및 "결합이 없는" 원자 제거
keep_idx = np.where(
(np.argmax(features, axis=1) != ATOM_DIM - 1)
& (np.sum(adjacency[:-1], axis=(0, 1)) != 0)
)[0]
features = features[keep_idx]
adjacency = adjacency[:, keep_idx, :][:, :, keep_idx]
# 분자에 원자 추가
for atom_type_idx in np.argmax(features, axis=1):
atom = Chem.Atom(atom_mapping[atom_type_idx])
_ = molecule.AddAtom(atom)
# [대칭] 인접 텐서의 상삼각형을 기반으로, 분자에 원자 간 결합 추가
(bonds_ij, atoms_i, atoms_j) = np.where(np.triu(adjacency) == 1)
for (bond_ij, atom_i, atom_j) in zip(bonds_ij, atoms_i, atoms_j):
if atom_i == atom_j or bond_ij == BOND_DIM - 1:
continue
bond_type = bond_mapping[bond_ij]
molecule.AddBond(int(atom_i), int(atom_j), bond_type)
# 분자를 정리합니다. 정리에 대한 추가 정보는
# https://www.rdkit.org/docs/RDKit_Book.html#molecular-sanitization 에서 확인하세요.
flag = Chem.SanitizeMol(molecule, catchErrors=True)
# 엄격하게 처리합니다. 정리에 실패하면, None을 반환합니다
if flag != Chem.SanitizeFlags.SANITIZE_NONE:
return None
return molecule
# 보조 함수 테스트
graph_to_molecule(smiles_to_graph(smiles))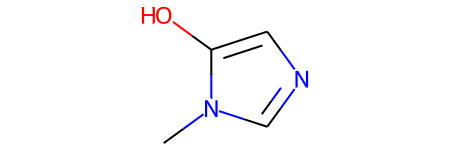
트레이닝 세트 생성
QM9 데이터셋의 일부만 사용하여, 트레이닝 시간을 절약합니다.
adjacency_tensor, feature_tensor = [], []
for smiles in data[::10]:
adjacency, features = smiles_to_graph(smiles)
adjacency_tensor.append(adjacency)
feature_tensor.append(features)
adjacency_tensor = np.array(adjacency_tensor)
feature_tensor = np.array(feature_tensor)
print("adjacency_tensor.shape =", adjacency_tensor.shape)
print("feature_tensor.shape =", feature_tensor.shape)결과
adjacency_tensor.shape = (13389, 5, 9, 9)
feature_tensor.shape = (13389, 9, 5)모델
이 예제의 목표는 WGAN-GP를 통해 생성자 네트워크와 판별자 네트워크를 구현하여, 작은 novel 분자(작은 그래프)를 생성할 수 있는 생성자 네트워크를 얻는 것입니다.
생성자 네트워크는 (배치 내의 각 예제에 대해) 벡터 z를 3차원 인접 텐서(A)와 2차원 특징 텐서(H)로 매핑할 수 있어야 합니다.
이를 위해, z는 먼저 완전 연결 네트워크를 통과하며, 그 출력은 두 개의 별도 완전 연결 네트워크로 전달됩니다.
각 완전 연결 네트워크는 배치 내 각 예제에 대해 tanh 활성화된 벡터를 출력한 후,
다차원 인접/특징 텐서와 일치하도록 reshape 및 소프트맥스를 수행합니다.
판별자 네트워크는 생성자 또는 트레이닝 데이터셋으로부터 온 그래프(A, H)를 입력으로 받게 됩니다.
이를 위해 그래프 상에서 연산할 수 있도록 그래프 컨볼루션 레이어를 구현해야 합니다.
이는 판별자 네트워크의 입력이 먼저 그래프 컨볼루션 레이어를 통과한 후,
평균 풀링 레이어와 몇 개의 완전 연결 레이어를 통과하게 됩니다.
최종 출력은 (배치 내 각 예제에 대해) 스칼라 값으로 나타나며,
이는 입력된 분자의 ‘진짜’ 또는 ‘가짜’ 여부를 나타냅니다.
그래프 생성자
def GraphGenerator(
dense_units, dropout_rate, latent_dim, adjacency_shape, feature_shape,
):
z = keras.layers.Input(shape=(LATENT_DIM,))
# 하나 이상의 완전 연결 레이어를 통해 전달
x = z
for units in dense_units:
x = keras.layers.Dense(units, activation="tanh")(x)
x = keras.layers.Dropout(dropout_rate)(x)
# 이전 레이어의 출력을 [연속적인] 인접 텐서로 매핑 (x_adjacency)
x_adjacency = keras.layers.Dense(tf.math.reduce_prod(adjacency_shape))(x)
x_adjacency = keras.layers.Reshape(adjacency_shape)(x_adjacency)
# 마지막 두 차원을 대칭화
x_adjacency = (x_adjacency + tf.transpose(x_adjacency, (0, 1, 3, 2))) / 2
x_adjacency = keras.layers.Softmax(axis=1)(x_adjacency)
# 이전 레이어의 출력을 [연속적인] 특징 텐서로 매핑 (x_features)
x_features = keras.layers.Dense(tf.math.reduce_prod(feature_shape))(x)
x_features = keras.layers.Reshape(feature_shape)(x_features)
x_features = keras.layers.Softmax(axis=2)(x_features)
return keras.Model(inputs=z, outputs=[x_adjacency, x_features], name="Generator")
generator = GraphGenerator(
dense_units=[128, 256, 512],
dropout_rate=0.2,
latent_dim=LATENT_DIM,
adjacency_shape=(BOND_DIM, NUM_ATOMS, NUM_ATOMS),
feature_shape=(NUM_ATOMS, ATOM_DIM),
)
generator.summary()결과
Model: "Generator"
__________________________________________________________________________________________________
Layer (type) Output Shape Param # Connected to
==================================================================================================
input_1 (InputLayer) [(None, 64)] 0
__________________________________________________________________________________________________
dense (Dense) (None, 128) 8320 input_1[0][0]
__________________________________________________________________________________________________
dropout (Dropout) (None, 128) 0 dense[0][0]
__________________________________________________________________________________________________
dense_1 (Dense) (None, 256) 33024 dropout[0][0]
__________________________________________________________________________________________________
dropout_1 (Dropout) (None, 256) 0 dense_1[0][0]
__________________________________________________________________________________________________
dense_2 (Dense) (None, 512) 131584 dropout_1[0][0]
__________________________________________________________________________________________________
dropout_2 (Dropout) (None, 512) 0 dense_2[0][0]
__________________________________________________________________________________________________
dense_3 (Dense) (None, 405) 207765 dropout_2[0][0]
__________________________________________________________________________________________________
reshape (Reshape) (None, 5, 9, 9) 0 dense_3[0][0]
__________________________________________________________________________________________________
tf.compat.v1.transpose (TFOpLam (None, 5, 9, 9) 0 reshape[0][0]
__________________________________________________________________________________________________
tf.__operators__.add (TFOpLambd (None, 5, 9, 9) 0 reshape[0][0]
tf.compat.v1.transpose[0][0]
__________________________________________________________________________________________________
dense_4 (Dense) (None, 45) 23085 dropout_2[0][0]
__________________________________________________________________________________________________
tf.math.truediv (TFOpLambda) (None, 5, 9, 9) 0 tf.__operators__.add[0][0]
__________________________________________________________________________________________________
reshape_1 (Reshape) (None, 9, 5) 0 dense_4[0][0]
__________________________________________________________________________________________________
softmax (Softmax) (None, 5, 9, 9) 0 tf.math.truediv[0][0]
__________________________________________________________________________________________________
softmax_1 (Softmax) (None, 9, 5) 0 reshape_1[0][0]
==================================================================================================
Total params: 403,778
Trainable params: 403,778
Non-trainable params: 0
__________________________________________________________________________________________________그래프 판별자
그래프 컨볼루션 레이어 (Graph convolutional layer). 관계 그래프 컨볼루션 레이어는 비선형적으로 변환된 이웃 정보 집계를 구현합니다. 이러한 레이어는 아래와 같이 정의할 수 있습니다:
$$ H^{l+1} = σ(D^{-1} @ A @ H^{l+1} @ W^{l}) $$
여기서
- $\sigma$는 비선형 변환을 의미하며(일반적으로 ReLU 활성화),
- $A$는 인접 텐서,
- $H^{l}$은 $l$번째 레이어에서의 특성 텐서,
- $D^{-1}$은 $A$의 역 대각 성분을 나타내며, (inverse diagonal degree tensor)
- $W^{l}$은 $l$번째 레이어에서 트레이닝 가능한 가중치 텐서를 의미합니다.
특히, 각 결합 유형(관계)마다, 대각선 성분은 각 원자에 연결된 결합의 개수를 나타냅니다. 이 튜토리얼에서는, 두 가지 이유로 인해 $D^{-1}$이 생략됩니다:
- (1) 생성자가 생성한 연속적인 인접 텐서에 대해 이 정규화를 적용하는 방법이 명확하지 않으며,
- (2) 정규화를 사용하지 않고도 WGAN의 성능이 양호하게 작동합니다.
또한, 원본 논문과 달리, 여기서는 자가 결합(self-loop)을 정의하지 않습니다. 이는 생성자가 자가 결합을 예측하도록 트레이닝하는 것을 방지하기 위함입니다.
class RelationalGraphConvLayer(keras.layers.Layer):
def __init__(
self,
units=128,
activation="relu",
use_bias=False,
kernel_initializer="glorot_uniform",
bias_initializer="zeros",
kernel_regularizer=None,
bias_regularizer=None,
**kwargs
):
super().__init__(**kwargs)
self.units = units
self.activation = keras.activations.get(activation)
self.use_bias = use_bias
self.kernel_initializer = keras.initializers.get(kernel_initializer)
self.bias_initializer = keras.initializers.get(bias_initializer)
self.kernel_regularizer = keras.regularizers.get(kernel_regularizer)
self.bias_regularizer = keras.regularizers.get(bias_regularizer)
def build(self, input_shape):
bond_dim = input_shape[0][1]
atom_dim = input_shape[1][2]
self.kernel = self.add_weight(
shape=(bond_dim, atom_dim, self.units),
initializer=self.kernel_initializer,
regularizer=self.kernel_regularizer,
trainable=True,
name="W",
dtype=tf.float32,
)
if self.use_bias:
self.bias = self.add_weight(
shape=(bond_dim, 1, self.units),
initializer=self.bias_initializer,
regularizer=self.bias_regularizer,
trainable=True,
name="b",
dtype=tf.float32,
)
self.built = True
def call(self, inputs, training=False):
adjacency, features = inputs
# 이웃으로부터 정보 집계
x = tf.matmul(adjacency, features[:, None, :, :])
# 선형 변환 적용
x = tf.matmul(x, self.kernel)
if self.use_bias:
x += self.bias
# 결합 유형 차원 축소
x_reduced = tf.reduce_sum(x, axis=1)
# 비선형 변환 적용
return self.activation(x_reduced)
def GraphDiscriminator(
gconv_units, dense_units, dropout_rate, adjacency_shape, feature_shape
):
adjacency = keras.layers.Input(shape=adjacency_shape)
features = keras.layers.Input(shape=feature_shape)
# 하나 이상의 그래프 컨볼루션 레이어를 통해 전파
features_transformed = features
for units in gconv_units:
features_transformed = RelationalGraphConvLayer(units)(
[adjacency, features_transformed]
)
# 분자의 2차원 표현을 1차원으로 축소
x = keras.layers.GlobalAveragePooling1D()(features_transformed)
# 하나 이상의 완전 연결 레이어를 통해 전파
for units in dense_units:
x = keras.layers.Dense(units, activation="relu")(x)
x = keras.layers.Dropout(dropout_rate)(x)
# 각 분자에 대해 입력된 분자의 '진짜' 여부를 나타내는 스칼라 값을 출력
x_out = keras.layers.Dense(1, dtype="float32")(x)
return keras.Model(inputs=[adjacency, features], outputs=x_out)
discriminator = GraphDiscriminator(
gconv_units=[128, 128, 128, 128],
dense_units=[512, 512],
dropout_rate=0.2,
adjacency_shape=(BOND_DIM, NUM_ATOMS, NUM_ATOMS),
feature_shape=(NUM_ATOMS, ATOM_DIM),
)
discriminator.summary()결과
Model: "model"
__________________________________________________________________________________________________
Layer (type) Output Shape Param # Connected to
==================================================================================================
input_2 (InputLayer) [(None, 5, 9, 9)] 0
__________________________________________________________________________________________________
input_3 (InputLayer) [(None, 9, 5)] 0
__________________________________________________________________________________________________
relational_graph_conv_layer (Re (None, 9, 128) 3200 input_2[0][0]
input_3[0][0]
__________________________________________________________________________________________________
relational_graph_conv_layer_1 ( (None, 9, 128) 81920 input_2[0][0]
relational_graph_conv_layer[0][0]
__________________________________________________________________________________________________
relational_graph_conv_layer_2 ( (None, 9, 128) 81920 input_2[0][0]
relational_graph_conv_layer_1[0][
__________________________________________________________________________________________________
relational_graph_conv_layer_3 ( (None, 9, 128) 81920 input_2[0][0]
relational_graph_conv_layer_2[0][
__________________________________________________________________________________________________
global_average_pooling1d (Globa (None, 128) 0 relational_graph_conv_layer_3[0][
__________________________________________________________________________________________________
dense_5 (Dense) (None, 512) 66048 global_average_pooling1d[0][0]
__________________________________________________________________________________________________
dropout_3 (Dropout) (None, 512) 0 dense_5[0][0]
__________________________________________________________________________________________________
dense_6 (Dense) (None, 512) 262656 dropout_3[0][0]
__________________________________________________________________________________________________
dropout_4 (Dropout) (None, 512) 0 dense_6[0][0]
__________________________________________________________________________________________________
dense_7 (Dense) (None, 1) 513 dropout_4[0][0]
==================================================================================================
Total params: 578,177
Trainable params: 578,177
Non-trainable params: 0
__________________________________________________________________________________________________WGAN-GP
class GraphWGAN(keras.Model):
def __init__(
self,
generator,
discriminator,
discriminator_steps=1,
generator_steps=1,
gp_weight=10,
**kwargs
):
super().__init__(**kwargs)
self.generator = generator
self.discriminator = discriminator
self.discriminator_steps = discriminator_steps
self.generator_steps = generator_steps
self.gp_weight = gp_weight
self.latent_dim = self.generator.input_shape[-1]
def compile(self, optimizer_generator, optimizer_discriminator, **kwargs):
super().compile(**kwargs)
self.optimizer_generator = optimizer_generator
self.optimizer_discriminator = optimizer_discriminator
self.metric_generator = keras.metrics.Mean(name="loss_gen")
self.metric_discriminator = keras.metrics.Mean(name="loss_dis")
def train_step(self, inputs):
if isinstance(inputs[0], tuple):
inputs = inputs[0]
graph_real = inputs
self.batch_size = tf.shape(inputs[0])[0]
# 하나 또는 그 이상의 스텝에 대해 판별자 트레이닝
for _ in range(self.discriminator_steps):
z = tf.random.normal((self.batch_size, self.latent_dim))
with tf.GradientTape() as tape:
graph_generated = self.generator(z, training=True)
loss = self._loss_discriminator(graph_real, graph_generated)
grads = tape.gradient(loss, self.discriminator.trainable_weights)
self.optimizer_discriminator.apply_gradients(
zip(grads, self.discriminator.trainable_weights)
)
self.metric_discriminator.update_state(loss)
# 하나 또는 그 이상의 스텝에 대해 생성자 트레이닝
for _ in range(self.generator_steps):
z = tf.random.normal((self.batch_size, self.latent_dim))
with tf.GradientTape() as tape:
graph_generated = self.generator(z, training=True)
loss = self._loss_generator(graph_generated)
grads = tape.gradient(loss, self.generator.trainable_weights)
self.optimizer_generator.apply_gradients(
zip(grads, self.generator.trainable_weights)
)
self.metric_generator.update_state(loss)
return {m.name: m.result() for m in self.metrics}
def _loss_discriminator(self, graph_real, graph_generated):
logits_real = self.discriminator(graph_real, training=True)
logits_generated = self.discriminator(graph_generated, training=True)
loss = tf.reduce_mean(logits_generated) - tf.reduce_mean(logits_real)
loss_gp = self._gradient_penalty(graph_real, graph_generated)
return loss + loss_gp * self.gp_weight
def _loss_generator(self, graph_generated):
logits_generated = self.discriminator(graph_generated, training=True)
return -tf.reduce_mean(logits_generated)
def _gradient_penalty(self, graph_real, graph_generated):
# 그래프 언팩
adjacency_real, features_real = graph_real
adjacency_generated, features_generated = graph_generated
# 그래프 보간(interpolated) 생성 (adjacency_interp 및 features_interp)
alpha = tf.random.uniform([self.batch_size])
alpha = tf.reshape(alpha, (self.batch_size, 1, 1, 1))
adjacency_interp = (adjacency_real * alpha) + (1 - alpha) * adjacency_generated
alpha = tf.reshape(alpha, (self.batch_size, 1, 1))
features_interp = (features_real * alpha) + (1 - alpha) * features_generated
# 보간된 그래프에 대한 로짓 계산
with tf.GradientTape() as tape:
tape.watch(adjacency_interp)
tape.watch(features_interp)
logits = self.discriminator(
[adjacency_interp, features_interp], training=True
)
# 보간된 그래프에 대한 그래디언트 계산
grads = tape.gradient(logits, [adjacency_interp, features_interp])
# 그래디언트 패널티 계산
grads_adjacency_penalty = (1 - tf.norm(grads[0], axis=1)) ** 2
grads_features_penalty = (1 - tf.norm(grads[1], axis=2)) ** 2
return tf.reduce_mean(
tf.reduce_mean(grads_adjacency_penalty, axis=(-2, -1))
+ tf.reduce_mean(grads_features_penalty, axis=(-1))
)모델 트레이닝
시간을 절약하기 위해(CPU에서 실행하는 경우), 모델을 10 에포크 동안만 트레이닝합니다.
wgan = GraphWGAN(generator, discriminator, discriminator_steps=1)
wgan.compile(
optimizer_generator=keras.optimizers.Adam(5e-4),
optimizer_discriminator=keras.optimizers.Adam(5e-4),
)
wgan.fit([adjacency_tensor, feature_tensor], epochs=10, batch_size=16)결과
Epoch 1/10
837/837 [==============================] - 197s 226ms/step - loss_gen: 2.4626 - loss_dis: -4.3158
Epoch 2/10
837/837 [==============================] - 188s 225ms/step - loss_gen: 1.2832 - loss_dis: -1.3941
Epoch 3/10
837/837 [==============================] - 199s 237ms/step - loss_gen: 0.6742 - loss_dis: -1.2663
Epoch 4/10
837/837 [==============================] - 187s 224ms/step - loss_gen: 0.5090 - loss_dis: -1.6628
Epoch 5/10
837/837 [==============================] - 187s 223ms/step - loss_gen: 0.3686 - loss_dis: -1.4759
Epoch 6/10
837/837 [==============================] - 199s 237ms/step - loss_gen: 0.6925 - loss_dis: -1.5122
Epoch 7/10
837/837 [==============================] - 194s 232ms/step - loss_gen: 0.3966 - loss_dis: -1.5041
Epoch 8/10
837/837 [==============================] - 195s 233ms/step - loss_gen: 0.3595 - loss_dis: -1.6277
Epoch 9/10
837/837 [==============================] - 194s 232ms/step - loss_gen: 0.5862 - loss_dis: -1.7277
Epoch 10/10
837/837 [==============================] - 185s 221ms/step - loss_gen: -0.1642 - loss_dis: -1.5273
<keras.callbacks.History at 0x7ff8daed3a90>생성자를 사용하여 새로운 분자를 샘플링
def sample(generator, batch_size):
z = tf.random.normal((batch_size, LATENT_DIM))
graph = generator.predict(z)
# one-hot 인코딩된 인접 텐서를 얻음
adjacency = tf.argmax(graph[0], axis=1)
adjacency = tf.one_hot(adjacency, depth=BOND_DIM, axis=1)
# 자기 연결을 제거 (self-loop 제거)
adjacency = tf.linalg.set_diag(adjacency, tf.zeros(tf.shape(adjacency)[:-1]))
# one-hot 인코딩된 특징 텐서를 얻음
features = tf.argmax(graph[1], axis=2)
features = tf.one_hot(features, depth=ATOM_DIM, axis=2)
return [
graph_to_molecule([adjacency[i].numpy(), features[i].numpy()])
for i in range(batch_size)
]
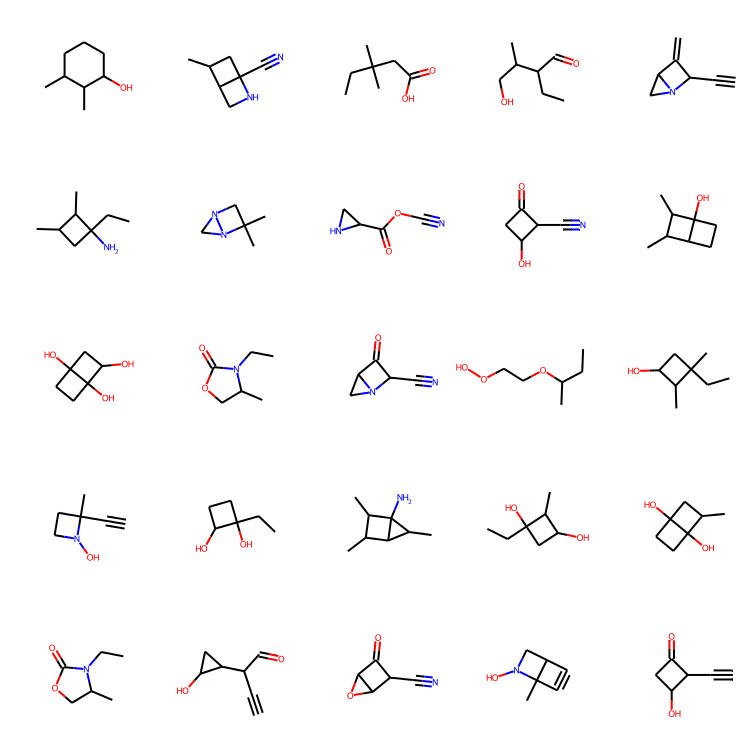
molecules = sample(wgan.generator, batch_size=48)
MolsToGridImage(
[m for m in molecules if m is not None][:25], molsPerRow=5, subImgSize=(150, 150)
)
마무리 생각
결과 검토: 10 에포크 동안의 트레이닝만으로도 꽤 그럴듯한 분자를 생성할 수 있었습니다! MolGAN 논문과 비교했을 때, 이번 튜토리얼에서 생성된 분자의 고유성이 상당히 높다는 점이 매우 고무적입니다!
학습한 내용과 앞으로의 전망: 이 튜토리얼에서는 분자 그래프를 생성하는 모델을 성공적으로 구현하였고, 이를 통해 새로운 분자를 생성할 수 있었습니다. 앞으로는, 기존 분자를 수정하는 생성 모델을 구현해보는 것도 흥미로울 것입니다. 예를 들어, 기존 분자의 용해도나 단백질 결합성을 최적화하는 방법을 연구할 수 있습니다. 이를 위해서는 재구성 손실(reconstruction loss)을 추가해야 할 가능성이 높으며, 이는 두 분자 그래프 간의 유사성을 쉽게 계산하는 방법이 없기 때문에 구현이 어려울 수 있습니다.
이 예제는 HuggingFace에서 확인 가능합니다.
| 트레이닝된 모델 | 데모 |
|---|---|
 |  |